LOS RESULTADOS DE LA COMPOSICIÓN GENÉTICA EN UN CONTEXTO GLOBAL – UNA COLABORACIÓN CON LA UNIVERSIDAD DE “TEXAS AT TYLER”.
Una de las principales características de ADNTRO es la descripción de la composición de ancestralidad de cada individuo (junto con la Metabolización de las Vitaminas, Deporte, Comportamiento, la Predisposición a las Enfermedades…) etc., pero todos los análisis que realizamos tienen como fuente de inspiración los mejores estudios publicados al respecto.
Esta metodología alternativa de análisis ancestral, está basada en una publicación de Nature (una de las revistas científicas más prestigiosas del mundo) y en la base de datos 1000 Genomes; a pesar de que esta metodología no nos permite ofrecer el desglose detallado por países que ofrecemos en nuestra calculadora de ancestros de ADNTRO, es un método muy robusto con el que podrás aprender como se analiza la ancestría de un individuo.
Para lograr la descripción de la composición de ancestralidad, utilizamos ‘pipelines’ computacionales (flujos de trabajo para “big data”- grandes conjuntos de datos – que utilizan la potencia computacional de Google Cloud) para comparar tu ADN con el de muchos otros individuos de todo el mundo, en este caso utilizando como punto de partida la base de datos 1000 Genome, que comprende 2.504 perfiles genéticos de individuos anónimos de 26 poblaciones mundiales diferentes (ver el apartado de información adicional que aparece más abajo para más detalles).
Basándonos en tu similitud genética, nuestros algoritmos informáticos hacen predicciones sobre qué parte de tu ascendencia procede de diferentes lugares del mundo y de cuáles.
Es importante situar los resultados en un contexto global. Algunas personas de todo el mundo tienen ancestros exclusivos de la región de la que proceden, pero muchas otras tienen ancestros mixtos que pueden reflejar patrones de migración humana en el pasado lejano o en épocas recientes (como tener un progenitor de un país diferente o incluso de un continente distinto).
ADNTRO ha colaborado con el Dr. Joshua Banta, profesor de Biología de la Universidad de Texas en Tyler, que estudia y publica sobre la genética de las poblaciones naturales, utilizando Big data para poner a tu disposición una herramienta visual que sitúa tus resultados genéticos en un contexto mundial.
Si nos detenemos un poco más en la metodología, queremos contarte que utilizamos un algoritmo de factorización de matrices no negativas (sNMF) implementado por la función ‘snmf’ del paquete LEA perteneciente al lenguaje de programación R. Este algoritmo estima las contribuciones al genoma de hipotéticos grupos ancestrales de cada individuo en el análisis, basándose en las similitudes/diferencias genéticas de las secuencias de ADN entre sí.
Para encontrar el número óptimo de grupos ancestrales (K), se simularon diferentes números de ancestros hipotéticos y se compararon las diferentes simulaciones para determinar cuál se ajustaba mejor a los datos con los que tratamos y, por tanto, qué número de grupos K debía utilizarse para esta nueva herramienta. Probamos diferentes números de grupos K, entre 5 y 12, utilizando el criterio de entropía de la función sNMF, que evalúa la calidad del ajuste del modelo estadístico a los datos mediante una técnica de validación cruzada (Ver información adicional (1)).
Se favorece el modelo con un determinado número de grupos K cuando se minimiza el criterio de entropía (Ver información adicional (2)).
Basándonos en estos criterios, encontramos que el número óptimo de grupos K es 5 (Figura 1).
Para conocer más detalles sobre los métodos, puedes consultar la información adicional situada al final del artículo).
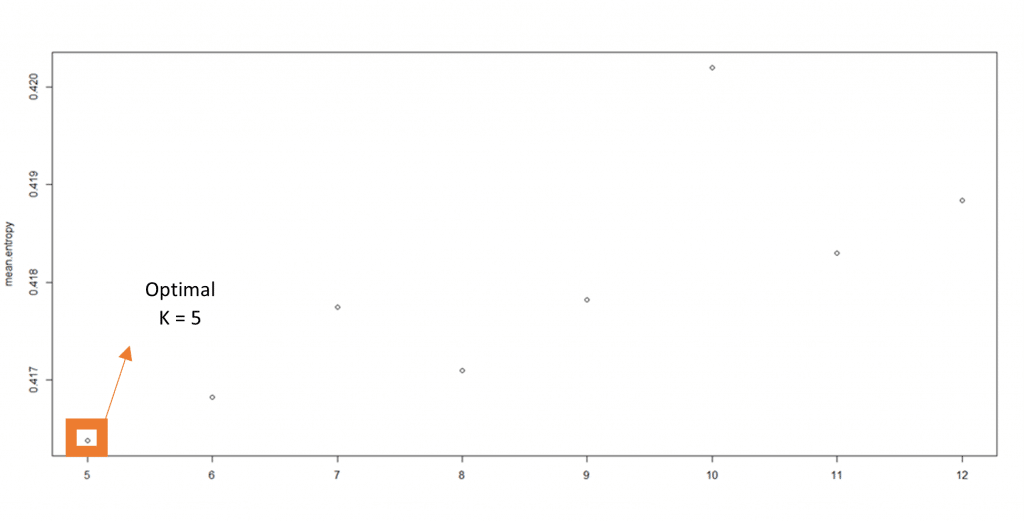
Figura 1. El número de grupos ancestrales es óptimo para un valor de cinco. La entropía se minimiza cuando el valor de K es cinco, siendo cinco el número que mejor se ajusta a nuestros datos.
TU COMPOSICIÓN DE ANCESTRALIDAD A NIVEL GEOGRÁFICO
Estos cinco grupos ancestrales pueden describirse visualmente como ancestros de África (representados en rojo), Amerindio (representados en verde), Eurasia Occidental (representados en azul clarito), Asia Oriental (representado en amarillo) y Asia del sur (representados en naranja).
Para obtener la contribución de dichos grupos ancestrales en tu ADN, ejecutamos el análisis con los datos de 1000 Genomes junto con tus datos para generar los resultados que incluyen tu ascendencia en un contexto mundial.
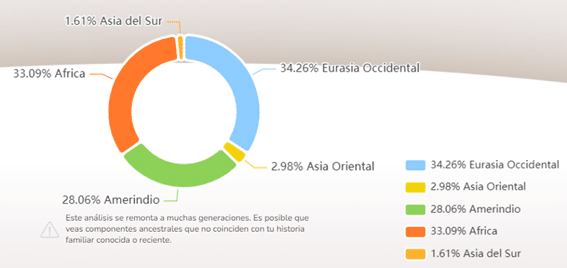
Figura 2. Diagrama en el que se desglosan los cinco componentes mayoritarios y el porcentaje de los mismos.
Con este gráfico podrás ver más claramente el patrón de los grupos ancestrales, es decir, la contribución de las diferentes ascendencias a tu ADN y al resto de las poblaciones de 1000G. Asimismo, podrás conocer la similitud que tienes con las distintas poblaciones de 1000G (Figura 3).
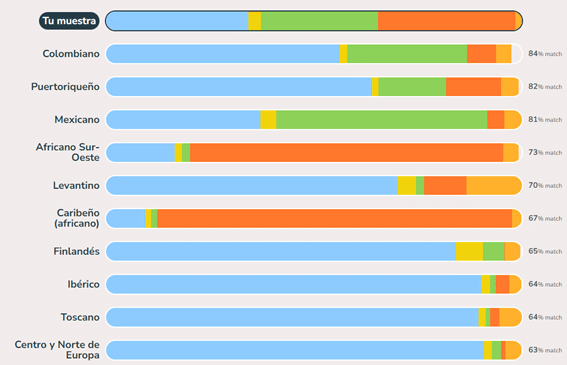
Figura 3. Diagrama en el que se desglosan los cinco componentes mayoritarios y se indica la similitud genética que tienes con las distintas poblaciones de 1000G.
Cada segmento multicolor representa a un componente principal. El individuo de este ejemplo tiene colores mixtos debido a pequeñas cantidades de ascendencia mixta. Curiosamente, los perfiles genéticos de los pueblos de América son los que mayor variabilidad presentan.
Hay muchos individuos que tienen una ascendencia sustancial asociada con Eurasia Occidental (azul clarito), así como contribuciones sustanciales asociadas con ancestros africanos (rojo). Esto refleja la compleja historia de los pueblos africanos con la trata de esclavos y con los pueblos de las Américas, donde los indígenas se mezclaron con los colonos europeos.
PATRONES ANCESTRALES
Si ya formas parte de la comunidad de ADNTRO, sabrás que nos gusta ir más allá, ser creativos y ofrecer a nuestros usuarios resultados innovadores. Por ese motivo hemos desarrollado un visor comparativo de patrones ancestrales basado en correlaciones. Utilizando los datos de 1000 Genomes y los tuyos hemos analizado cuán de parecido y cuán de distinto es tu ADN con el “típico” ADN de las 26 poblaciones diferentes de 1000 Genomes.
Esta es una nueva forma de ver tu ancestría. Las zonas del gráfico (Figura 4) que ves más altas son las poblaciones con las cuales compartes un gran número de marcadores específicos presentes en dicha población. Por el contrario, las zonas más bajas de la gráfica representan aquellas poblaciones con las que presentas una menor similitud.
Desde ADNTRO nos gusta ofrecer nuevos ángulos e innovar, siempre teniendo en mente la importancia de entender tu ADN como un todo; esto es lo que te permite obtener tu patrón ancestral y compararlo con distintos patrones ancestrales a nivel mundial. Algo sin precedentes en el mundo de los test genéticos directos al consumidor a día de hoy.
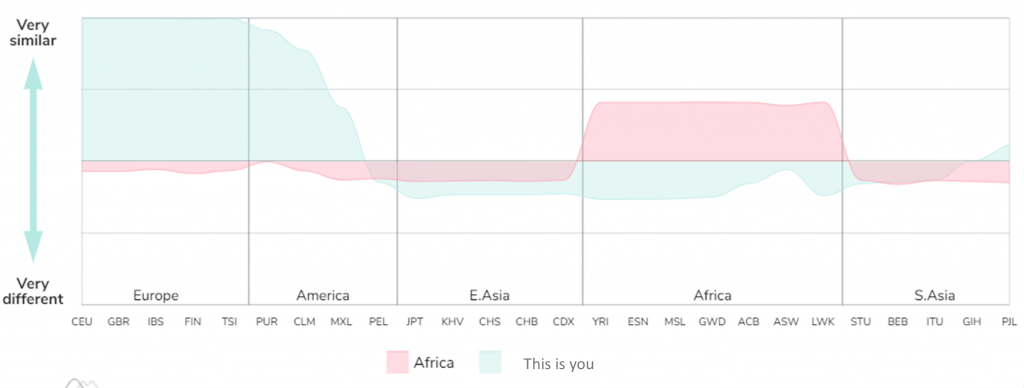
Figura 4. Comparativa entre el patrón del “típico” africano con el tuyo. Este gráfico es un ejemplo en el cual utilizamos una muestra europea. Sin embargo, el patrón cambiará acorde a tu información genética. Del mismo modo, podrás cambiar las poblaciones de referencia contra las que te quieras comparar.
El hecho de que tu patrón se parezca mucho a poblaciones similares a ti es muy normal. Dos individuos europeos, por ejemplo, tienen un patrón muy similar si lo miramos en su conjunto y únicamente presenta pequeñas diferencias que son las que merecen la pena explorar (las diferencias son tan pequeñas que necesitaríamos exagerarlas en una escala logarítmica).
Es importante que tengas en mente que el hecho de que tu patrón tenga una similitud muy alta con otra persona no significa que estéis relacionados a nivel familiar, sino que tu patrón ancestral es muy similar porque presentáis ancestros comunes.
¡Pero esto no acaba aquí! Te diremos también con qué poblaciones de 1000 Genomes te asemejas más y con cuáles presentas una mayor diferencia. Adicionalmente, si ya eres cliente de ADNTRO podrás subir tu patrón junto con una población de referencia y un alias, para poder compartir con otros patrones del mundo no incluidos en la base de datos de 1000G.
¿Te gusta esta herramienta? ¿Tienes curiosidad por conocer tu patrón ancestral en un contexto global? ¿Quieres poder compararlo con otros? ¡Entra ADNTRO 😊, compra tu test de adn ancestros o sube tu “Raw” y descúbrelo!
INFORMACIÓN ADICIONAL
(1) Puedes consultar la página 926 del artículo, en la primera columna, cerca del final de la página, en la sección «Análisis de la estructura de la población»)
(2) Puedes consultar la página 927 del artículo, al final.
Más detalles sobre los métodos: Tanto para los datasets de 1000 Genomes como tus propios resultados, filtramos nuestra base de datos de polimorfismos (SNPs) para quedarnos únicamente con aquellos que son bialélicos, con un MAF ≥ 0.05 y con un espaciamiento por lo menos de 2kb.
¿Qué poblaciones incluye el proyecto 1000 Genomes?:
- African Ancestry in SW USA [ASW]
- African Caribbean in Barbados [ACB]
- Bengali in Bangladesh [BEB]
- British From England and Scotland [GBR]
- Chinese Dai in Xishuangbanna, China [CDX]
- Northern Europeans from Utah and TSI means Tuscans from Italy [CEU]
- Colombian in Medellín, Colombia [CLM]
- Esan in Nigeria [ESN]
- Finnish in Finland [FIN]
- Gujarati Indians in Houston, Texas, USA []
- Han Chinese in Beijing, China [CHB]
- Han Chinese South [CHS]
- Iberian Populations in Spain [IBS]
- Indian Telugu in the U.K. [ITU]
- Japanese in Tokyo, Japan [JPT]
- Kinh in Ho Chi Minh City, Vietnam [KHV]
- Luhya in Webuye, Kenya [LWK]
- Mende in Sierra Leone [MSL]
- Mexican Ancestry in Los Angeles CA USA [MXL]
- Peruvian in Lima Peru [PEL]
- Puerto Rican in Puerto Rico [PUR]
- Punjabi in Lahore, Pakistan [PJL]
- Sri Lankan Tamil in the UK [STU]
- Toscani in Italia [TSI]
- Yoruba in Ibadan, Nigeria [YRI]
- Gambian in Western Division – Mandinka [GWD]